세 번째 프로젝트 순서
1. 식물 병충해 자료 파일분류
2. CNN 모델링
3. 이미지화
4. CNN, AlexNet, VGG-16 모델 평가
예전에 네이버 블로그를 하듯이 글을 자유롭게 쓰고 싶은데, 혹시나 틀릴까 하는 마음에 쉽게 글이 작성이 되지 않는다. 너무나 쉽게 생각을 한 것이 아닌가 싶기도 하지만.. 그래도 팀프로젝트 5개 + 현재하고 있는 파이널 프로젝트 관련된 글을 작성하려고 한다.
이번에는 실패했던 시각화에 대한 내용이라 너무 관심 있게 보지는 말아주셨으면 한다. (부끄러우니까)
CNN 모델링을 했을 때 정확도가 90퍼센트 이상이 나왔지만 이를 어떻게 하면 더 개선시킬 수 있을까에 대한 고민을 많이 했다. 그러다가 생각한 것이 학습에 필요한 데이터는 전체 사진이 아닌 식물의 사진만 중요하지 않을까? 그래서 배경을 제외하고 학습을 해보기로 했다.
-배경과 식물 분리하는 mask
-sharpen 값을 줘서 병충해가 있는 데이터는 조금 더 부각이 되게

여러 예제들을 찾아보다가 이거다! 하는 예제가 있어서 처음에 단순히 따라하는 식으로 해봤다! 이건 그냥 너무 쉽게 되겠는 걸?




결과는 참담했다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
|
#배경과 분리하기 위해서 hsv 값 조정
def create_mask_for_plant(image):
image_hsv = cv2.cvtColor(image, cv2.COLOR_BGR2HSV)
sensitivity = 30
lower_hsv = np.array([60 - sensitivity, 100, 50])
upper_hsv = np.array([60 + sensitivity, 255, 255])
mask = cv2.inRange(image_hsv, lower_hsv, upper_hsv)
kernel = cv2.getStructuringElement(cv2.MORPH_ELLIPSE, (11,11))
mask = cv2.morphologyEx(mask, cv2.MORPH_CLOSE, kernel)
return mask
#비트연산 and 모든 색이 같을 때만 색 표현
def segment_plant(image):
mask = create_mask_for_plant(image)
output = cv2.bitwise_and(image, image, mask = mask)
return output
#샤픈, 특징을 조금더 부각시키게
def sharpen_image(image):
image_blurred = cv2.GaussianBlur(image, (0, 0), 3)
image_sharp = cv2.addWeighted(image, 1.5, image_blurred, -0.5, 0)
return image_sharp
|
cs |

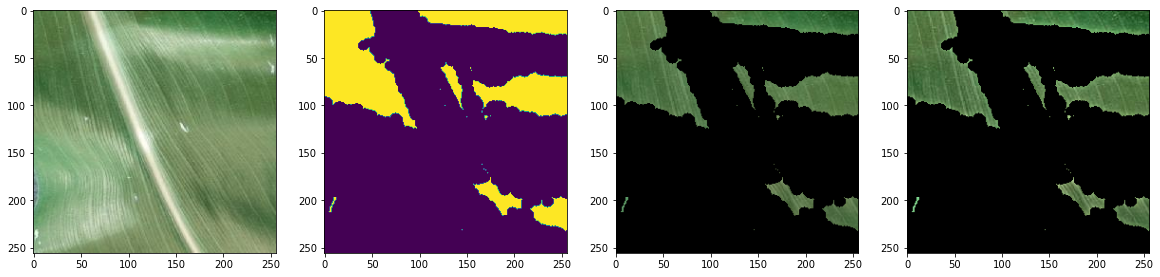
sensitivity를 어떻게 하느냐에 따라서 결과값이 많이 차이가 났다. 위의 경우가 1을 뒀을 때고, 아래의 경우가 30일 때 실행을 했는데 단점이 배경 없이 잎만 찍은 데이터의 경우 분간이 제대로 되지가 않았다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
|
%matplotlib inline
import os
import matplotlib
import matplotlib.pyplot as plt
import pandas as pd
import cv2
import numpy as np
from glob import glob
import seaborn as sns
image = cv2.imread('8_9_2.jpg')
image_mask = create_mask_for_plant(image)
image_segmented = segment_plant(image)
image_sharpen = sharpen_image(image_segmented)
fig, axs = plt.subplots(1, 4, figsize=(20, 20))
axs[0].imshow(image)
axs[1].imshow(image_mask)
axs[2].imshow(image_segmented)
axs[3].imshow(image_sharpen)
|
cs |

그래도 이전보다는 배경과 잎의 경계가 뚜렷하게 나눠지는 것을 확인할 수 있었다. 그런데도 문제가 되는 부분은 잎을 나눈데 있어서 hsv 값을 비슷하게 판단해서인지? 식물의 잎도 같이 사라지는 마술을 보여줬다. 병충해가 있어서 저렇게 빵꾸가 뚤렸다고 생각하면 좋겠지만, 병충해가 있는 부분은 중간 아래부분이다.
라이트룸이나 사진 편집을 할 때, 어떤 식으로 특정 색만 색깔을 남길까 생각을 해봤을 때.. hsv 전체 색 공간이 아닌 말 그대로 특정색만 추출하면 되는데 왜 이생각을 못 했을까 하고 무릎을 탁 쳤다. 그래서 초록색을 가장 잘 나타낼 수 있는 값들을 조절해봤다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
import numpy as np
import cv2
img_color = cv2.imread('4_7_5.jpg')
img_hsv = cv2.cvtColor(img_color, cv2.COLOR_BGR2HSV) # cvtColor 함수를 이용하여 hsv 색공간으로 변환
lower_green = (0, 30, 30)
upper_green = (80, 255, 255)
img_mask = cv2.inRange(img_hsv, lower_green, upper_green) #hsv 색공간 전체가 아닌 지정한 범위 내의
img_result = cv2.bitwise_and(img_color, img_color, mask = img_mask) # 바이너리 이미지를 마스크로 사용하여 원본이미지에서 범위값에 해당하는 부분을 획득
fig, axs = plt.subplots(1, 3, figsize=(20, 20))
axs[0].imshow(img_color)
axs[1].imshow(img_mask)
axs[2].imshow(img_result)
|
cs |
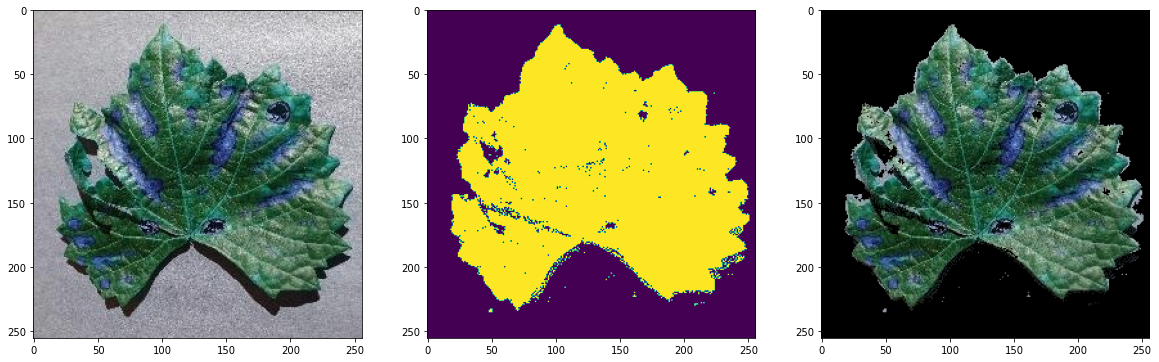
이정도면 나름 선방한 결과였다. 하지만 이 이미지들을 다 다시 저장시키고, 식물마다 톤이 다르고, 사진 찍은 곳 조명이 달라서 lower_green과 upper_green을 일일이 다 조정해야 한다는 치명적인 단점이 있어서 실패를 했다.
그래도 어떤 식으로 사진과 배경을 분리하고, 샤픈을 줘서 CNN이 사진의 특징을 학습할 때 조금 더 도움을 줄 수 있지 않을까 하는 생각이 든다.
'도전하자. 프로젝트' 카테고리의 다른 글
| 4-1 파이썬 팀프로젝트 CNN 상품 카테고리 분류 intro (셀리니움 vs BS4) (0) | 2021.08.10 |
|---|---|
| 3-4 파이썬 팀프로젝트 - CNN, AlexNet, VGG-16 모델 평가 (0) | 2021.08.09 |
| 3-2 파이썬 팀프로젝트 CNN 모델링 - 인공지능, 머신러닝, 딥러닝 뭔데? (0) | 2021.08.01 |
| 3-1 파이썬 팀프로젝트 CNN 식물 병충해 분류 (0) | 2021.07.31 |
| 2-4 파이썬 팀프로젝트 : matplotlib, smtplib 메일 자동화 (0) | 2021.07.19 |